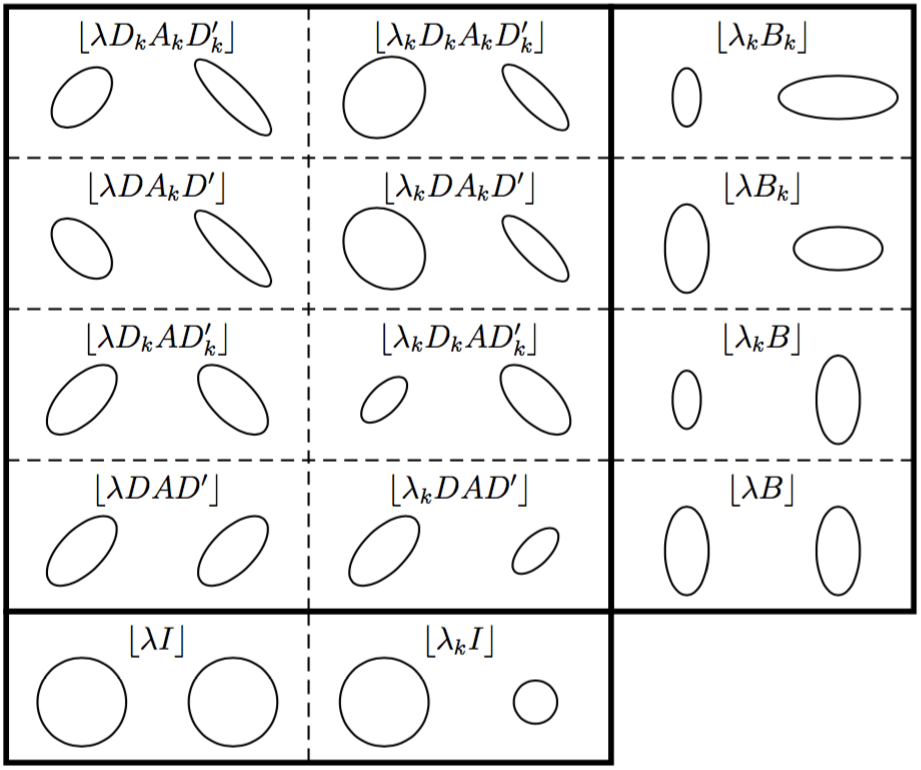
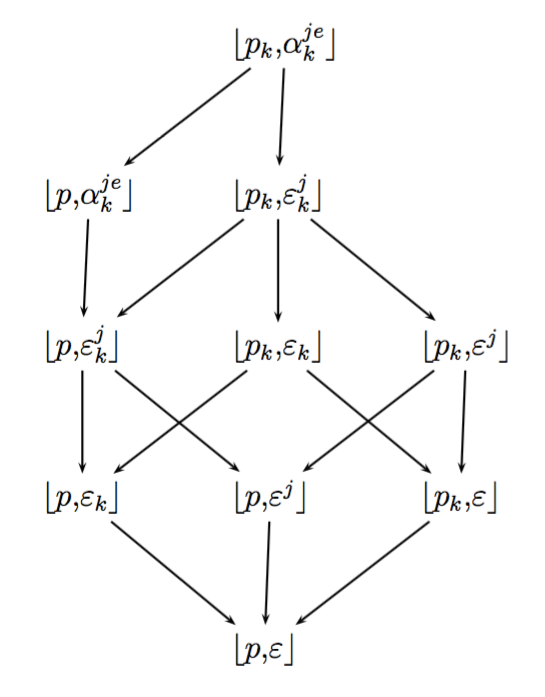
Plan
- Problème de classification
- Partition
- Hiérarchie
- Modèles de mélange
- Algorithmes EM et CEM
- Nombre de classes
- Critères de choix
- Applications
- avec code
R
- avec code
Librairies R utilisées
library(mclust) library(knitr) library(Rmixmod) library(ggplot2) library(reshape2) library(NbClust) library(gridExtra)